Nature Biotechnology, NeoDisc:蛋白质基因组学新抗原发现流程推动个性化癌症免疫治疗
一、研究背景及目的
研究背景:个性化肿瘤抗原的发现至关重要,其中识别由 I 类和 II 类人类白细胞抗原(HLA-I 和 HLA-II)呈现在患者癌细胞表面且能被自体 T 细胞识别的肽段,对于癌症疫苗、T 细胞过继转移以及各种 T 细胞靶向分子的开发意义重大。基于全基因组测序(WGS)或全外显子组测序(WES)以及 RNA 测序(RNAseq)的突变抗原(新抗原)预测方法常被应用于转化研究和临床试验。近年来,质谱(MS)与蛋白质基因组学相结合用于识别 HLA 结合肽,推动了对癌症中天然加工和呈递的各类靶标的探索,涵盖新抗原、肿瘤相关抗原和肿瘤特异性抗原(TAA 和 TSA)、肿瘤病毒以及推定的非蛋白质编码转录本翻译的肽段。然而,这些抗原的识别过程极为繁琐,现有的临床流程并不支持免疫肽组学,且仅限于预测的新抗原。
研究目的:本研究旨在引入一个名为 NeoDisc 的 “端到端” 临床抗原发现蛋白质基因组学流程。NeoDisc 整合了最先进的公开及内部软件,用于从基因组学、转录组学以及基于 MS 的免疫肽组学中识别免疫原性肿瘤特异性 HLA-I 和 HLA-II 抗原,并运用基于规则和机器学习(ML)的工具进行预测和优先排序。该流程旨在评估肿瘤异质性以及抗原加工呈递机制(APPM)的功能,将其应用于转化癌症研究中的个性化抗原发现以及癌症疫苗设计的临床实施。
二、研究方法
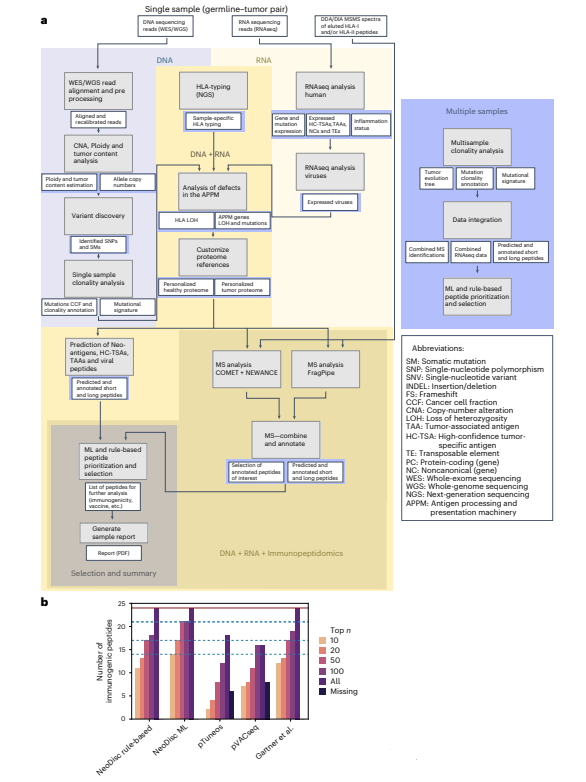
NeoDisc 流程整合了来自不同方面的数据,包括基因组、转录组和免疫肽组学数据。主要方法有:
三、研究结果
研究人员运用 NeoDisc 流程对来自七位患有不同癌症的患者的肿瘤样本进行深入分析,结果显示:
NeoDisc 优于现有的新抗原优先排序流程:NeoDisc 的 ML 优先排序算法相较于 pTuneos、pVACtools 和 Gartner 等现有工具,能够更准确地对免疫原性新抗原进行优先排序。
NeoDisc 能够从多个来源识别免疫原性肿瘤特异性抗原:NeoDisc 成功地从多种来源(包括突变、TSA、病毒元素和非经典转录本)中检测并优先考虑了免疫原性 TSA,证明了其多组学数据整合的能力。
NeoDisc 揭示了与 APPM 缺陷相关的异质性抗原景观:NeoDisc 能够识别影响异质性肿瘤抗原景观的细胞抗原呈递机制缺陷,如 HLA LOH 和 B2M 突变。这些信息对于理解肿瘤免疫逃逸机制以及开发更有效的免疫治疗策略至关重要。
NeoDisc 能够设计个性化疫苗:NeoDisc 提供了三种操作模式(默认、敏感和面板),允许用户根据肿瘤含量和突变负荷调整分析参数。此外,NeoDisc 可以设计最大限度地覆盖高质量预测的 HLA-I 和 HLA-II 新抗原的长序列,并支持基于肽的疫苗中 N 端和 C 端的调整。
四、研究结论
NeoDisc 是一种先进的蛋白质基因组学流程,能够整合多组学数据,用于识别、预测和优先排序癌细胞上呈递的免疫原性抗原肽。它为个性化抗原发现和新抗原癌症疫苗设计提供了基于规则和机器学习的方法,并揭示了与 APPM 缺陷相关的异质性肿瘤抗原景观。NeoDisc 为转化研究和推进个性化癌症免疫治疗提供了强大的工具。
研究背景:个性化肿瘤抗原的发现至关重要,其中识别由 I 类和 II 类人类白细胞抗原(HLA-I 和 HLA-II)呈现在患者癌细胞表面且能被自体 T 细胞识别的肽段,对于癌症疫苗、T 细胞过继转移以及各种 T 细胞靶向分子的开发意义重大。基于全基因组测序(WGS)或全外显子组测序(WES)以及 RNA 测序(RNAseq)的突变抗原(新抗原)预测方法常被应用于转化研究和临床试验。近年来,质谱(MS)与蛋白质基因组学相结合用于识别 HLA 结合肽,推动了对癌症中天然加工和呈递的各类靶标的探索,涵盖新抗原、肿瘤相关抗原和肿瘤特异性抗原(TAA 和 TSA)、肿瘤病毒以及推定的非蛋白质编码转录本翻译的肽段。然而,这些抗原的识别过程极为繁琐,现有的临床流程并不支持免疫肽组学,且仅限于预测的新抗原。
研究目的:本研究旨在引入一个名为 NeoDisc 的 “端到端” 临床抗原发现蛋白质基因组学流程。NeoDisc 整合了最先进的公开及内部软件,用于从基因组学、转录组学以及基于 MS 的免疫肽组学中识别免疫原性肿瘤特异性 HLA-I 和 HLA-II 抗原,并运用基于规则和机器学习(ML)的工具进行预测和优先排序。该流程旨在评估肿瘤异质性以及抗原加工呈递机制(APPM)的功能,将其应用于转化癌症研究中的个性化抗原发现以及癌症疫苗设计的临床实施。
二、研究方法
NeoDisc 流程整合了来自不同方面的数据,包括基因组、转录组和免疫肽组学数据。主要方法有:
- 基因组数据分析:利用 WES 或 WGS 数据进行样本特异性变异表征、肿瘤含量估计以及拷贝数变异(CNV)和体细胞突变(SM)的识别。
- 转录组数据分析:使用 RNAseq 数据进行人类基因和 SM 表达量化,并估计 T 细胞炎症。未映射的 RNAseq reads 用于病毒感染识别和病毒基因表达量化。
- 免疫肽组学数据分析:将免疫肽组学数据与个性化蛋白质组参考数据库进行比对,以识别天然呈递的抗原肽。
- HLA 分型和 APPM 分析:从种系和肿瘤 WES 以及 RNAseq 数据中获取 HLA 分型。识别并突出显示肿瘤 APPM 中的缺陷以及 HLA 杂合性缺失(LOH)。
- 抗原预测和优先排序:采用基于规则和 ML 的方法对来自不同来源的肿瘤特异性和免疫原性抗原(包括新抗原、病毒抗原、高可信度肿瘤特异性抗原和肿瘤特异性非经典抗原)进行识别、预测和优先排序。
- 个性化疫苗设计:NeoDisc 支持基于规则和机器学习方法进行个性化抗原发现和新抗原癌症疫苗设计。
三、研究结果
研究人员运用 NeoDisc 流程对来自七位患有不同癌症的患者的肿瘤样本进行深入分析,结果显示:
NeoDisc 优于现有的新抗原优先排序流程:NeoDisc 的 ML 优先排序算法相较于 pTuneos、pVACtools 和 Gartner 等现有工具,能够更准确地对免疫原性新抗原进行优先排序。
NeoDisc 能够从多个来源识别免疫原性肿瘤特异性抗原:NeoDisc 成功地从多种来源(包括突变、TSA、病毒元素和非经典转录本)中检测并优先考虑了免疫原性 TSA,证明了其多组学数据整合的能力。
NeoDisc 揭示了与 APPM 缺陷相关的异质性抗原景观:NeoDisc 能够识别影响异质性肿瘤抗原景观的细胞抗原呈递机制缺陷,如 HLA LOH 和 B2M 突变。这些信息对于理解肿瘤免疫逃逸机制以及开发更有效的免疫治疗策略至关重要。
NeoDisc 能够设计个性化疫苗:NeoDisc 提供了三种操作模式(默认、敏感和面板),允许用户根据肿瘤含量和突变负荷调整分析参数。此外,NeoDisc 可以设计最大限度地覆盖高质量预测的 HLA-I 和 HLA-II 新抗原的长序列,并支持基于肽的疫苗中 N 端和 C 端的调整。
四、研究结论
NeoDisc 是一种先进的蛋白质基因组学流程,能够整合多组学数据,用于识别、预测和优先排序癌细胞上呈递的免疫原性抗原肽。它为个性化抗原发现和新抗原癌症疫苗设计提供了基于规则和机器学习的方法,并揭示了与 APPM 缺陷相关的异质性肿瘤抗原景观。NeoDisc 为转化研究和推进个性化癌症免疫治疗提供了强大的工具。