iCAGES:助力癌症个体化治疗的大数据分析工具

前言:iCAGES——世界上第一个无需生物信息基础即可上手的癌症基因组学分析工具,帮助临床医生快速分析和鉴定癌症患者所携带的驱动基因变异,并选择针对性治疗方案。
来自哥伦比亚大学医学研究中心(Columbia University Medical Center, CUMC)的王凯教授及其团队发布了一个旨在帮助癌症个体化治疗的大数据分析工具,通过鉴定患者的驱动基因变异和快速寻找相应的药物治疗方案,让医生在面对疾病大数据时化繁为简,优化诊疗方案。在所有同类分析工具当中,这是第一个具备简洁友好界面的专业分析工具,使得临床工作者不需要具备丰富的生物信息经验即可使用。这项研究于2016年12月22日发表于Genome Medicine杂志。
在分析结果方面,与现有的其它同类分析工具相比,这个被称为iCAGES的新软件从随计抽选的一组包含驱动、非驱动基因的候选基因中正确鉴定驱动基因的成功率高达77%——相比之下,其他相似的软件仅有51%的正确率。
大部分癌症的发生,都起因于体细胞遗传突变或其它变异的累积——在过去若干年研究中,与癌症发病机制有关的致病变异分析已在大规模患者样本中深入研究。然而,科研成果应用到临床却并不容易——癌症驱动基因具有明显的个体差异性,不同患者携带不同的驱动基因变异,而临床工作者恰恰缺少有效分析工具来分析和鉴定不同患者个体的驱动基因变异。“另一方面,面对数百种可能有效的药物,即使知道了患者携带哪种基因变异,医生也头疼应该如何才能制定合理治疗方案。”来自CUMC的基因组医学研究所临床信息学负责人、生物医学信息学教授王凯教授一直关注着临床医生在基因组学方面的挑战和困惑。
为了解决这一难题,王凯教授和他的团队开发了这个名为integrated CAncer GEnome Score(iCAGES)的分析工具:首先对患者的全基因组数据和其肿瘤组织的基因数据进行比较分析,寻找可能导致癌症发生的变异;其次,将潜在致癌变异与已知致癌基因变异数据库进行对比,通过生物信息分析、机器学习技术等方式来优化对比过程,最终鉴定最有可能的癌症驱动基因变异;在此基础上,iCAGES从所有FDA批准的、或正在临床试验阶段的癌症药物治疗手段中寻找最有可能匹配患者驱动基因变异的治疗方案。从一开始的基因组数据分析到最终找到可行治疗方案,iCAGES整个分析流程仅需约30分钟——相比之下,传统分析手段往往需要多个繁杂步骤和参数的人工设置,并且耗时数周才能得出最终结论。
王凯教授使用iCAGES对一例肺癌患者的基因组数据进行了回顾性分析。从129个潜在驱动基因变异中,iCAGES最终选定了ARAF这个基因,并由此在122个可能匹配的治疗方案中选择了索拉非尼(sorafenib)。虽然其主治医生也得到了同样的结论,但整个过程却费时费力,包括邀请肺癌专家会诊。“索拉非尼用药后,患者有了明显好转。”王凯教授说,“值得注意的是,FDA尚未正式批准索拉非尼用于此类适应症。这意味着iCAGES也有助于寻找新治疗手段、或者开拓现有FDA批准药物的非适应症用药。”
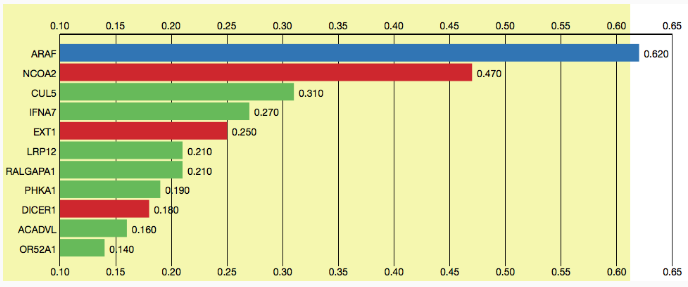
图1. iCAGES对一名肺癌患者的基因组数据进行了回顾性分析,从而找到了其所携带的癌症驱动基因ARAF
除此之外,从各类癌症患者案例的实际分析结果来看,iCAGES无论是癌症驱动基因变异鉴定还是寻找针对性治疗方案,都优于同类分析工具的表现。
“我们希望iCAGES能够帮助临床医生更全面地受益于基因组学和疾病大数据领域的快速发展,从而令癌症个性化治疗更进一步。”王凯教授提到他们这项研究的初衷时说道。
来源:哥伦比亚大学医学研究中心